
|
|||||
|
|
| 菌群16S rRNA基因测序的聚类分析方法研究进展 | |
| 引用本文: | 尚家起, 刘婧, 李佳颖, 等. 菌群16S rRNA基因测序的聚类分析方法研究进展[J]. 中国微生态学杂志, 2023, 35(7): 864-868. doi: 10.13381/j.cnki.cjm.202307023 |
| 作者姓名: | 尚家起 刘婧 李佳颖 邵雷 |
| 作者单位: | 1. 上海理工大学健康科学与工程学院,上海 200093; 2. 上海健康医学院协同科研中心; 3. 上海中医药大学 |
| 摘 要: | 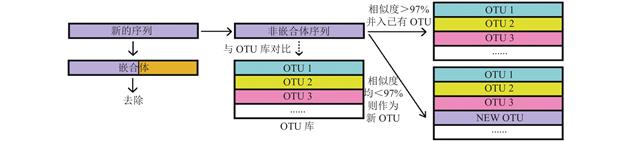 16S核糖体RNA(16S rRNA)基因测序是微生物分析的重要手段。16S rRNA基因测序的原始数据复杂,存在许多误差,分析前一般需要先进行序列质量控制,即对数据进行去噪、去冗余和去嵌合,最终将质量控制后的数据分为可操作分类单元(OTU)或ASV,在OTU或ASV基础上再进行菌群的各种分析。  |
| 关 键 词: | 菌群分析 16S rRNA基因测序 聚类方法 菌群分析软件 |
| 收稿时间: | 2021-11-03 |
| 修稿时间: | 2022-05-18 |
| 点击此处可从《中国微生态学杂志》浏览原始摘要信息 | |
| 点击此处可从《中国微生态学杂志》下载免费的PDF全文 | |
