| 染色质互作相关转录因子的挖掘及功能分析 |
| |
| 作者姓名: | 王琪 丁砚书 耿宝宝 聂玉敏 |
| |
| 作者单位: | 南京医科大学生物医学工程与信息学院,生物信息学系,南京 210029,南京医科大学生物医学工程与信息学院,生物信息学系,南京 210029,南京医科大学生物医学工程与信息学院,生物信息学系,南京 210029,南京医科大学生物医学工程与信息学院,生物信息学系,南京 210029 |
| |
| 基金项目: | 江苏省自然科学基金青年基金项目(BK20161026),江苏省高校自然科学研究面上项目(16KJB180022)和南京医科大学科技发展基金重点项目(2015NJMUZD003)资助. |
| |
| 摘 要: | 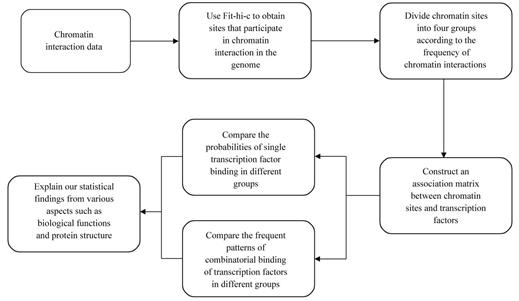
染色质互作是真核生物基因组组装的基础,并且在调控真核基因细胞特异性表达中发挥重要作用.染色质互作的发生与特定的蛋白质有关,目前已经发现CTCF (CCCTC binding facor,转录阻抑物)和黏连蛋白与染色质互作相关,然而并不清楚是否还有其他蛋白质参与染色质互作.我们将整合高通量染色体构象捕获(Hi-C)和染色质免疫沉淀-测序(ChIP-seq)数据,在GM12878和K562细胞系中挖掘与染色质互作相关的转录因子,并对发现的转录因子做功能分析.
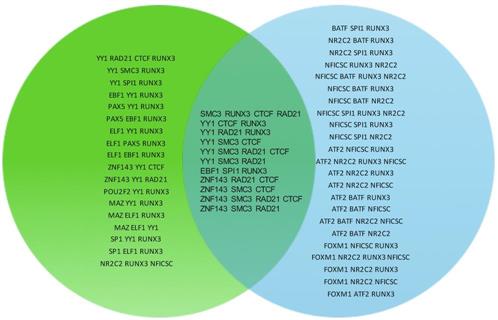
我们在频繁发生互作的染色质位点中发现RUNX3、SPI1等转录因子也可能参与染色质互作.另外,通过FP-growth的数据挖掘方法还发现多个转录因子可能协同作用参与染色质互作.研究结果将为染色质互作相关实验的开展提供先验知识.
|
| 关 键 词: | 染色质互作 转录因子 FP-growth Hi-C |
| 收稿时间: | 2018-11-23 |
| 修稿时间: | 2019-02-20 |
| 本文献已被 CNKI 等数据库收录! |
| 点击此处可从《生物化学与生物物理进展》浏览原始摘要信息 |
|
点击此处可从《生物化学与生物物理进展》下载免费的PDF全文 |
|



