| 基于注意力机制的RNA碱基关联图预测方法 |
| |
| 作者姓名: | 曹一航 黄强 |
| |
| 作者单位: | 复旦大学生命科学学院,上海 200438,复旦大学生命科学学院,上海 200438 |
| |
| 基金项目: | 国家重大科技专项“重大新药开发”课题(2018ZX09J18112)和国家自然科学基金(31971377)资助项目。 |
| |
| 摘 要: | 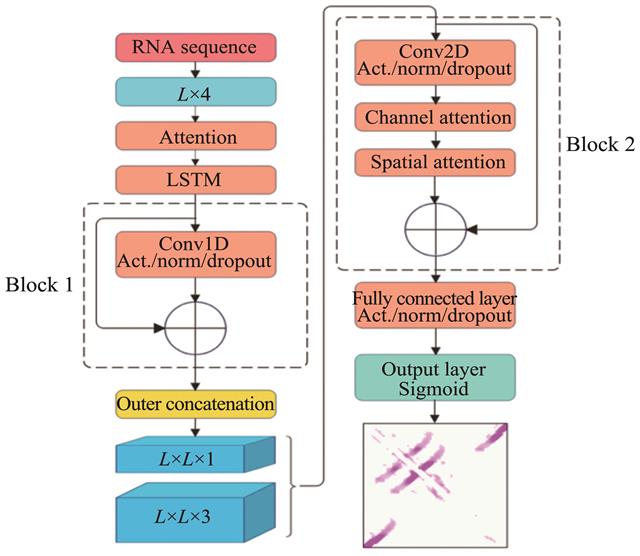
目的 长链非编码RNA在遗传、代谢和基因表达调控等方面发挥着重要作用。然而,传统的实验方法解析RNA的三级结构耗时长、费用高且操作要求高。此外,通过计算方法来预测RNA的三级结构在近十年来无突破性进展。因此,需要提出新的预测算法来准确的预测RNA的三级结构。所以,本文发展可以用于提高RNA三级结构预测准确性的碱基关联图预测方法。
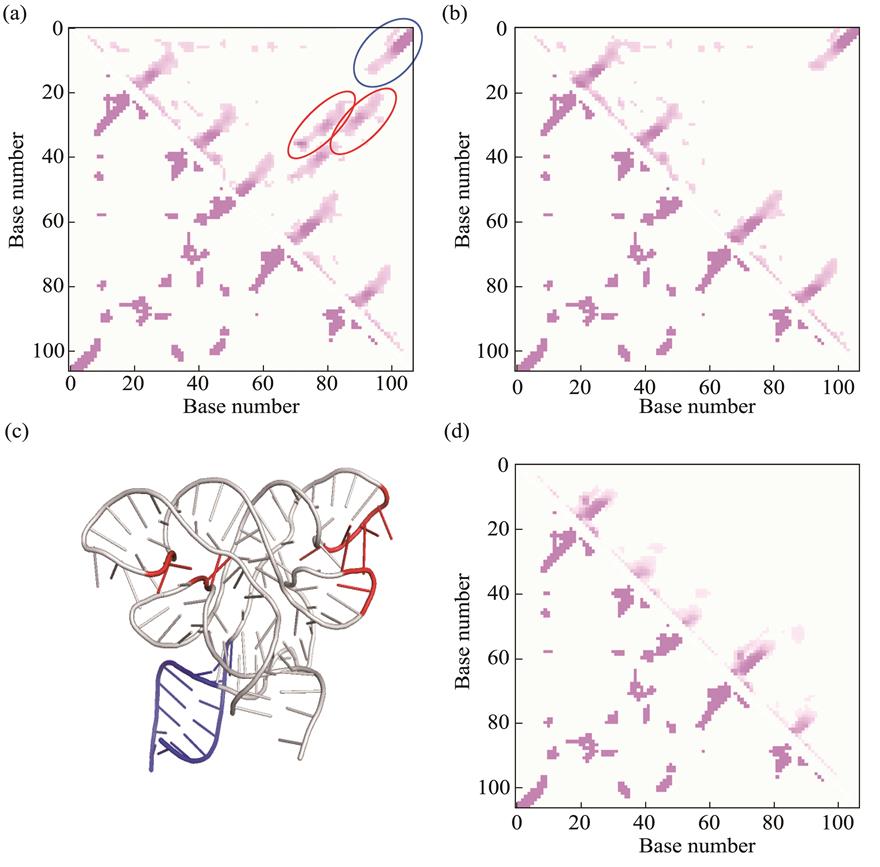
方法 为了利用RNA理化特征信息,本文应用多层全卷积神经网络和循环神经网络的深度学习算法来预测RNA碱基间的接触概率,并通过注意力机制处理RNA序列中碱基间相互依赖的特征。结果 通过多层神经网络与注意力机制结合,本文方法能够有效得到RNA特征值中局部和全局的信息,提高了模型的鲁棒性和泛化能力。检验计算表明,所提出模型对序列长度L的4种标准(L/10、L/5、L/2、L)碱基关联图的预测准确率分别达到0.84、0.82、0.82和0.75。结论 基于注意力机制的深度学习预测算法能够提高RNA碱基关联图预测的准确率,从而帮助RNA三级结构的预测。
|
| 关 键 词: | 深度学习 RNA碱基关联图 结构预测 注意力机制 |
| 收稿时间: | 2022-05-26 |
| 修稿时间: | 2022-07-11 |
|
| 点击此处可从《生物化学与生物物理进展》浏览原始摘要信息 |
|
点击此处可从《生物化学与生物物理进展》下载免费的PDF全文 |
|



