| 基于双层BiGRU网络的哺乳动物组织m6A甲基化位点预测 |
| |
| 作者姓名: | 李慧敏 陈鹏辉 唐轶 徐权峰 胡梦 王煜 |
| |
| 作者单位: | 云南民族大学数学与计算机科学学院,昆明 650504,云南民族大学数学与计算机科学学院,昆明 650504,云南民族大学数学与计算机科学学院,昆明 650504,云南民族大学数学与计算机科学学院,昆明 650504,云南民族大学数学与计算机科学学院,昆明 650504,云南民族大学数学与计算机科学学院,昆明 650504 |
| |
| 基金项目: | 国家自然科学基金(61866040)资助项目。 |
| |
| 摘 要: | 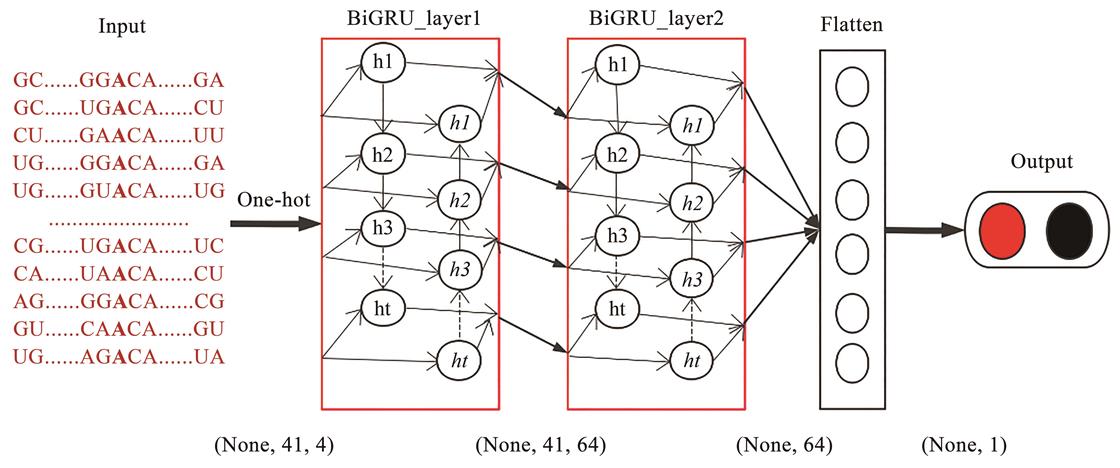
目的 N6-甲基化腺苷(N6-methyladenosine,m6A)是RNA中最常见、最丰富的化学修饰,在很多生物过程中发挥着重要作用。目前已经发展了一些预测m6A甲基化位点的计算方法。然而,这些方法在针对不同物种或不同组织时,缺乏稳健性。为了提升对不同组织中m6A甲基化位点预测的稳健性,本文提出一种能结合序列反向信息来提取数据更高级特征的双层双向门控循环单元(bidirectional gated recurrent unit,BiGRU)网络模型。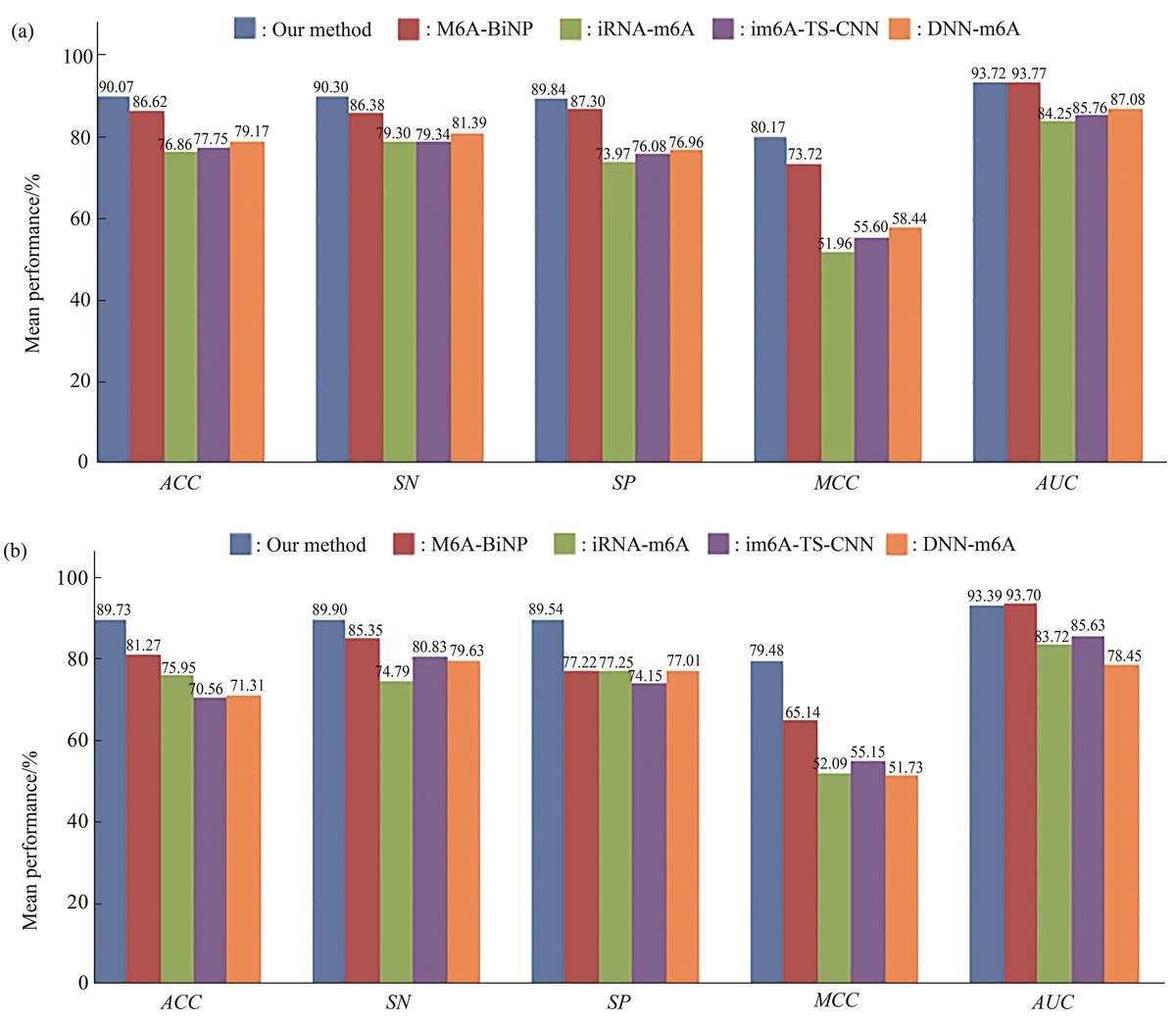
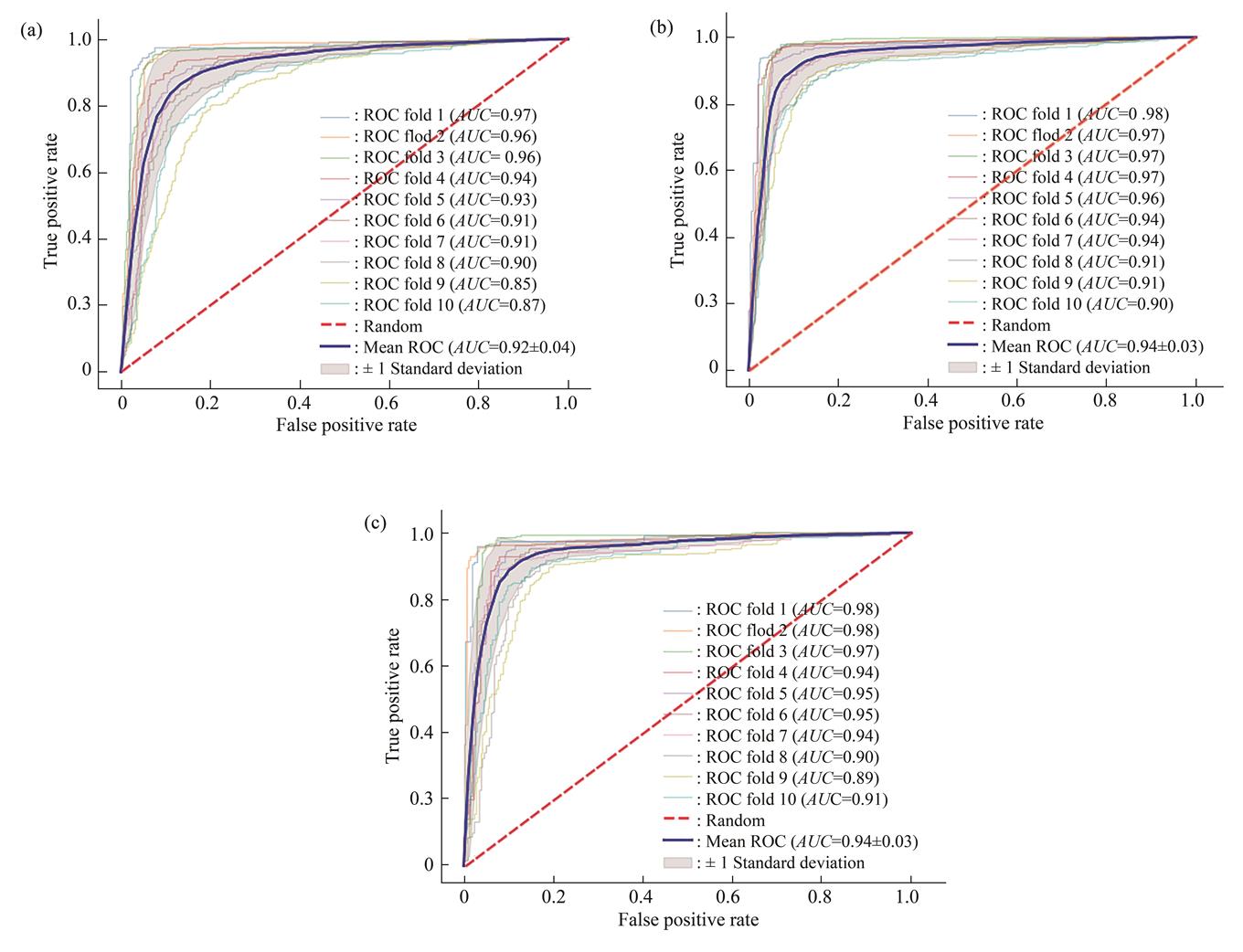
方法 本文选取具有代表性的哺乳动物组织m6A甲基化位点数据集作为训练数据,通过对模型网络、网络结构、层数和优化器等进行搭配,构建双层BiGRU网络。结果 将模型应用于人类、小鼠和大鼠共11个组织的m6A甲基化位点预测上,并与其他方法在这11个组织上的预测能力进行了全面的比较。结果表明,本文构建的模型平均预测接受者操作特征曲线下面积(area under the receiver operating characteristic curve,AUC)达到93.72%,与目前最好的预测方法持平,而预测准确率(accuracy,ACC)、敏感性(sensitivity,SN)、特异性(specificity,SP)和马修斯相关系数(Matthews correlation coefficient,MCC)分别为90.07%、90.30%、89.84%和80.17%,均高于目前的m6A甲基化位点预测方法。结论 和已有研究方法相比,本文方法对11个哺乳动物组织的m6A甲基化位点的预测准确性均达到最高,说明本文方法具有较好的泛化能力。
|
| 关 键 词: | N6-甲基化腺苷位点 双向门控循环单元 碱基序列 深度学习 |
| 收稿时间: | 2023-01-09 |
| 修稿时间: | 2023-11-06 |
|
| 点击此处可从《生物化学与生物物理进展》浏览原始摘要信息 |
|
点击此处可从《生物化学与生物物理进展》下载免费的PDF全文 |
|



