| 基于卷积神经网络和循环神经网络的环形RNA剪接位点识别研究 |
| |
| 作者姓名: | 孙凯 魏庆功 臧超禹 孙如轩 姜丹 孙晓勇 |
| |
| 作者单位: | 山东农业大学信息科学与工程学院农业大数据研究中心,泰安 271000,山东农业大学信息科学与工程学院农业大数据研究中心,泰安 271000,山东农业大学信息科学与工程学院农业大数据研究中心,泰安 271000,山东农业大学信息科学与工程学院农业大数据研究中心,泰安 271000,山东农业大学信息科学与工程学院农业大数据研究中心,泰安 271000,山东农业大学信息科学与工程学院农业大数据研究中心,泰安 271000 |
| |
| 基金项目: | 国家自然科学基金(32070684, 31571306)资助项目. |
| |
| 摘 要: | 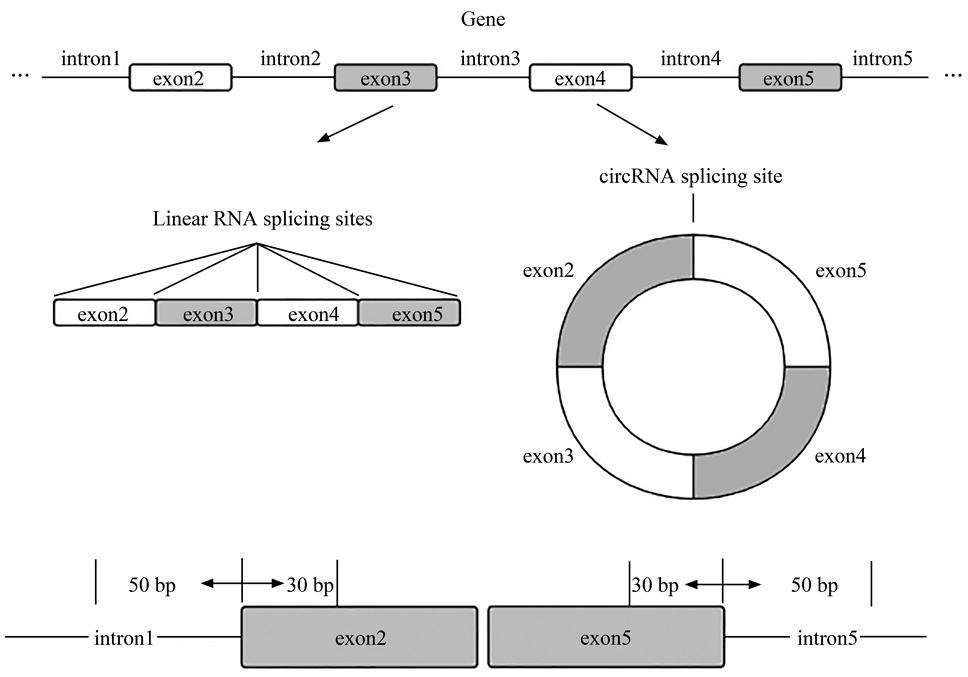
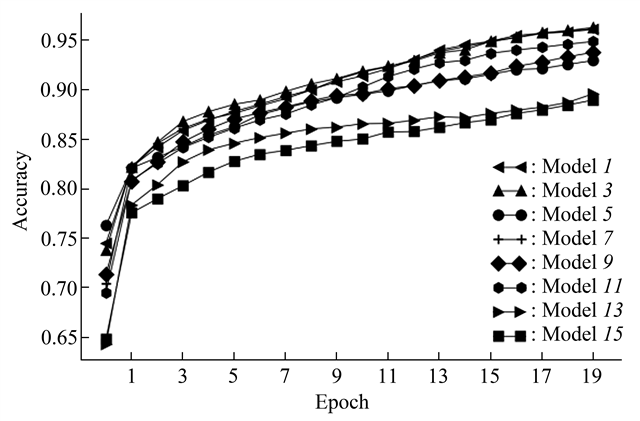
本文提出了一种基于卷积神经网络和循环神经网络的深度学习模型,通过分析基因组序列数据,识别人基因组中环形RNA剪接位点.首先,根据预处理后的核苷酸序列,设计了2种网络深度、8种卷积核大小和3种长短期记忆(long short term memory,LSTM)参数,共8组16个模型;其次,进一步针对池化层进行均值池化和最大池化的测试,并加入GC含量提高模型的预测能力;最后,对已经实验验证过的人类精浆中环形RNA进行了预测.结果表明,卷积核尺寸为32×4、深度为1、LSTM参数为32的模型识别率最高,在训练集上为0.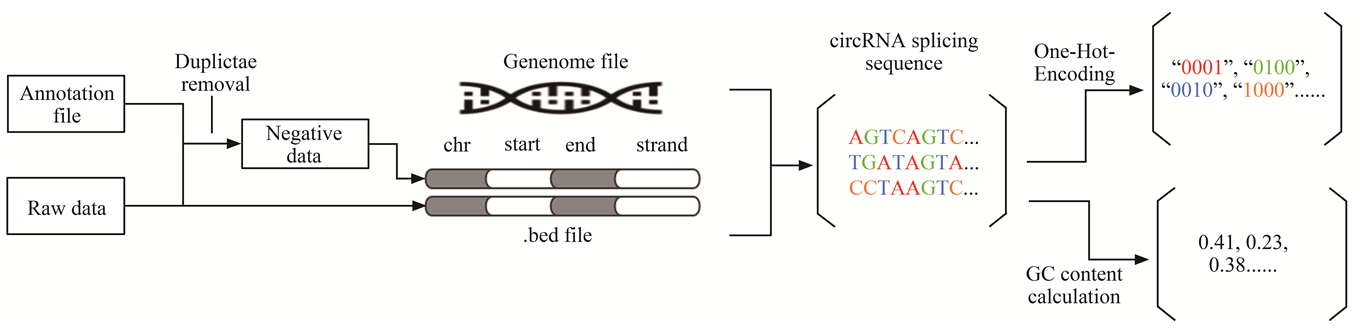
9824,在测试数据集上准确率为0.95,并且在实验验证数据上的正确识别率为83%.该模型在人的环形RNA剪接位点识别方面具有较好的性能.
|
| 关 键 词: | 深度学习 卷积神经网络 循环神经网络 环形RNA 剪接位点 |
| 收稿时间: | 2020-08-18 |
| 修稿时间: | 2020-09-10 |
| 本文献已被 CNKI 维普 等数据库收录! |
| 点击此处可从《生物化学与生物物理进展》浏览原始摘要信息 |
|
点击此处可从《生物化学与生物物理进展》下载全文 |
|



